2022-12-15 | 科研部 南科大靳文菲团队建立单细胞polyA测序新方法scPloyA-seq
近日,南方科技大学生命科学学院副教授靳文菲团队联合复旦大学教授倪挺团队在PNAS在线发表了题为“Comprehensive mapping of alternative polyadenylation site usage and its dynamics at single cell resolution”的科研论文。该研究建立了单细胞多聚腺苷酸测序技术(scPolyA-seq)和相关分析流程。利用该技术分析了polyA位点的使用在单细胞水平的动态变化,发现polyA位点的使用伴随着细胞周期变化呈现出几种不同的变化模式;发现敲除 MSL1或SCCPDH等基因的polyA位点能加快或减缓细胞周期。这些结果这预示着polyA位点使用的转换在细胞周期调控中发挥重要作用。
在真核生物中,PolyA (多聚腺苷酸) 通常是位于成熟mRNA末端共价链结的 50-250个腺苷酸残基。PolyA参与了mRNA成熟、mRNA出核、mRNA稳定、mRNA降解和mRNA的细胞定位等过程,在转录后基因调控中发挥着重要作用。一个基因的mRNA前体因选择性使用不同polyA 位点产生多种mRNA异构体,极大的丰富了mRNA产物的多样性(图1A)。polyA位点的选择性使用通过影响蛋白编码、或3′UTR区域的基序(motif), 甚至蛋白的功能、丰度、细胞内亚定位等,进而影响生物学过程。但常规单细胞转录组测序主要关注基因表达量而忽略了对polyA位点的捕获。这样导致我们对polyA位点使用的认识仍然主要停留在基于大量细胞的数据,对 polyA位点使用在细胞间的差异知之甚少。
该研究通过改进SMART-seq2 来特异性捕获mRNA的3′端来获得更多携带polyA位点的读长(reads)。结合 C1 单细胞自动制备系统对多个细胞系进行scPolyA-seq并建立了 scPolyA-seq的分析流程。相对于10x Genomics单细胞转录组方法(scRNA-seq),scPolyA-seq获得的读长数、基因数、携带polyA位点的读长占比和数目都有大幅提升(图1)。
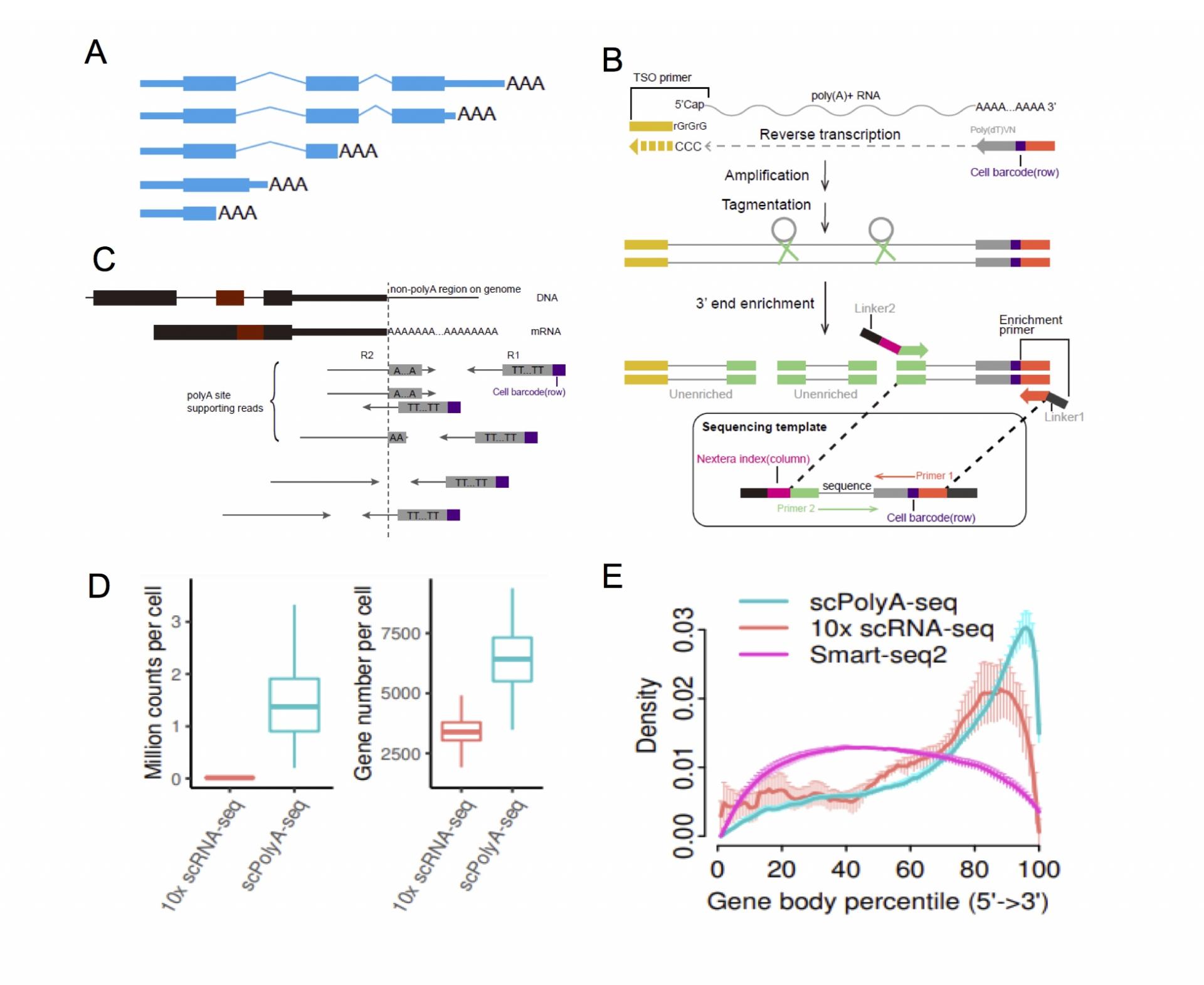
图1. scPolyA-seq方法及其优势。(A) 基因的多个polyA位点极大的丰富了转录本的多样性。(B) scPolyA-seq 示意图。(C) 通过携带polyA位点的读长来分析polyA位点的使用。(D)scPolyA-seq与 10x单细胞转录组的获得读长书和基因数比较。(E) scPolyA-seq 的reads更多地集中在mRNA的3′端。
通过分析多个细胞系的 scPolyA-seq,研究团队发现大量细胞数据(bulk data)中使用了多个polyA位点的基因倾向于在单细胞层面仅使用一个polyA位置。有趣的是,对 scPolyA-seq数据分析发现polyA位点使用高度变异的基因列表中富集了细胞周期相关基因。研究团队进一步发现细胞周期同步化后polyA位点使用转换的基因列表富集了细胞周期相关基因,但细胞周期同步化后基因表达量变化的基因列表却没有富集细胞周期相关基因。研究团队根据细胞周期基因在细胞内的表达水平估推算了每个细胞的细胞周期状态。通过对相邻细胞周期之间polyA 位点使用的变化分析,发现这些随细胞周期改变 polyA 位点使用的基因呈现出六种动态模式,每种模式的基因列表富集了不同类型的功能基因,即每种模式有特定功能。研究团队敲除MSL1 和 SCCPDH 等基因的一个polyA位点加快或减缓了细胞周期。这些结果预示着polyA位点使用的转换在细胞周期调控中发挥重要作用。
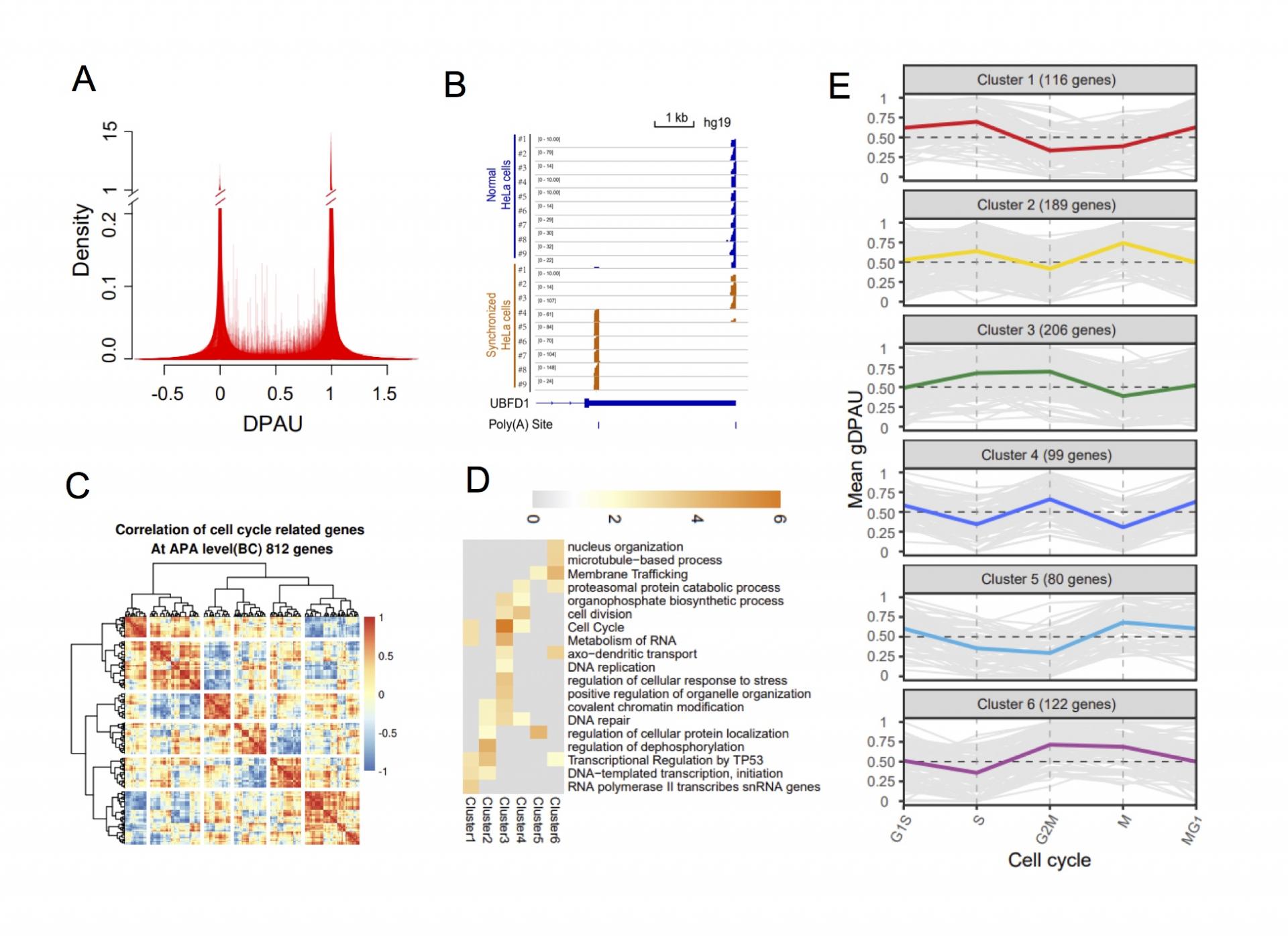
图2. polyA位点使用的特点及其与细胞周期的关系。(A)一个细胞倾向于只使用一个polyA位点。(B)UBFD1的polyA位点使用在细胞周期同步化前后的变化。(C)细胞周期过程中polyA位点使用变化的基因聚成了6个簇。(D)每簇基因列表富集了不同类型的功能基因。(E)每簇基因的polyA位点使用呈现一种动态模式。
在南方科技大学访学的郑州大学博士生王军亮和复旦大学博士生陈炜为该论文共同第一作者。靳文菲为论文最后通讯作者,共同通讯作者包括复旦大学教授倪挺、南方科技大学生命科学学院研究助理教授洪旎、郑州大学教授祁元明 。南方科技大学生命科学学院副教授饶枫和原助理教授仲寒冰参与了该项工作。
该研究得到了国家重点研发计划、国家自然科学基金委员会和深圳市科技创新委员会的资助。
论文链接:https://www.pnas.org/doi/10.1073/pnas.2113504119
供稿:生命科学学院
通讯员:付文卿
主图:丘妍
编辑:朱增光